Tutorial para iniciantes do Piximi (Portugues)#
Segmentação e classificação sem instalação no navegador#
Beth Cimini, Le Liu, Esteban Miglietta, Paula Llanos, Nodar Gogoberidze
Instituto Broad do MIT e Harvard, Cambridge, MA.
Informações básicas:#
O que é Piximi?#
Piximi é uma ferramenta moderna de análise de imagens sem programação que utiliza aprendizado profundo. Implementado como um aplicativo web em https://piximi.app/, o Piximi não requer instalação e pode ser acessado por qualquer navegador moderno. Sua arquitetura exclusiva para clientes preserva a segurança dos dados do pesquisador, executando toda a computação localmente.
O Piximi é interoperável com ferramentas e fluxos de trabalho existentes, suportando importação e exportação de dados e formatos de modelos comuns. A interface intuitiva e o fácil acesso ao Piximi permitem que pesquisadores obtenham insights sobre imagens em apenas alguns minutos. O Piximi visa levar a análise de imagens com aprendizado profundo a uma comunidade mais ampla, eliminando barreiras.
* exceto as segmentações usando Cellpose, que são enviadas para um servidor remoto (com a permissão do usuário).
Funcionalidades principais: Anotador, Segmentador, Classificador, Medições.
Objetivo do exercício#
Neste exercício, você se familiarizará com as principais funcionalidades do Piximi: anotação, segmentação, classificação, mensuração e visualização, e o utilizará para analisar um conjunto de imagens de um experimento de translocação. O objetivo deste experimento é determinar a menor dose efetiva de Wortmannin necessária para induzir a localização nuclear de FOXO1A marcada com GFP (Figura 21). Você segmentará as imagens usando um dos modelos de aprendizado profundo disponíveis no Piximi, verificará e selecionará a segmentação e, em seguida, treinará um classificador de imagens para classificar as células individuais como tendo “GFP nuclear”, “GFP citoplasmática” ou “sem GFP”. Por fim, você fará medições e as plotará para responder à pergunta biológica.
Contexto do experimento#
Neste experimento, pesquisadores obtiveram imagens de células U2OS de osteossarcoma (câncer ósseo) fixadas expressando uma proteína de fusão FOXO1A-GFP e coraram DAPI para marcar os núcleos. FOXO1 é um fator de transcrição que desempenha um papel fundamental na regulação da gliconeogênese e glicogenólise por meio da sinalização da insulina. FOXO1A transita dinamicamente entre o citoplasma e o núcleo em resposta a vários estímulos. A wortmanina, um inibidor da PI3K, pode bloquear a exportação nuclear, resultando no acúmulo de FOXO1A no núcleo.
Materiais necessários para este exercício#
Os materiais necessários para este exercício podem ser baixados de: PiximiTutorial. O arquivo “Piximi Translocation Tutorial RGB.zip” contém um projeto Piximi, incluindo todas as imagens, já rotuladas com o tratamento correspondente (concentração de Wortmannin ou Controle). Baixe este arquivo, mas NÃO o descompacte!
Instruções do exercício#
Leia os passos abaixo e siga as instruções onde indicado. Os passos em que você precisa encontrar uma solução estão marcados com 🔴 PARA FAZER.
1. Carregue o projeto Piximi#
🔴 PARA FAZER
Inicie o Piximi acessando: https://piximi.app/
Carregue o projeto de exemplo: Clique em “Abrir” - “Projeto” - “Projeto do Zip”, como mostrado na figura 22, para carregar um arquivo de projeto para este tutorial do Zip. Você também pode alterar o nome do projeto no painel superior esquerdo, como “Exercício Piximi”. Conforme ele é carregado, você pode ver a progressão no logotipo
 no canto superior esquerdo.
no canto superior esquerdo.
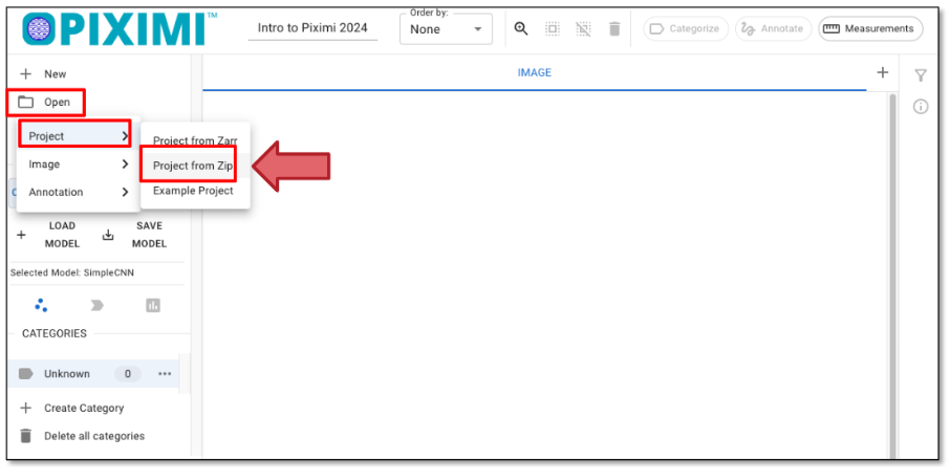
Fig. 41 Carregando um arquivo de projeto.#
2. Verifique as imagens carregadas e explore a interface do Piximi#
Estas 17 imagens representam tratamentos com Wortmannin em oito concentrações diferentes (expressas em nM), bem como tratamentos controles (0 nM). Observe que o canal DAPI (Núcleos) é mostrado em magenta e que o canal GFP (FOXOA1) é mostrado em verde.
Ao passar o mouse sobre a imagem, rótulos coloridos são exibidos no canto esquerdo das imagens. Essas anotações são dos metadados do arquivo compactado que acabamos de enviar. Neste tutorial, os diferentes rótulos coloridos indicam a concentração de Wortmannin, enquanto os números representam o número de imagens em cada categoria.
Opcionalmente, você pode anotar as imagens manualmente clicando em “+ Categoria”, inserindo seu rótulo e, em seguida, selecionando a imagem clicando nas imagens e anotando as imagens selecionadas clicando em “Categorizar”. Neste tutorial, pularemos esta etapa, pois os rótulos já foram carregados no início.
3. Segmentar Células - descubra as células a partir do fundo#
🔴 PARA FAZER
Para iniciar a previsão em todas as imagens, clique em “Selecionar Todas as Imagens” no painel superior, conforme mostrado na Figura 23.
Altere a Tarefa de Aprendizagem para “SEGMENTAÇÃO” (Figura 24, Seta 1).
Clique em “+ CARREGAR MODELO” (Seta 2) e a janela será exibida, permitindo que você escolha um modelo pré-treinado (Seta 3). Para o exercício de hoje, selecione “Cellpose” (Seta 4). Mais informações sobre o modelo suportado podem ser encontradas aqui.
Clique em “Abrir Modelo de Segmentação” (Seta 5) para carregar seu modelo e selecioná-lo. Por fim, clique em “Prever Modelo” (Seta 5). Você verá o progresso da previsão exibido no canto superior esquerdo, abaixo do logotipo da Piximi
 .
.A segmentação levará alguns minutos para ser concluída.
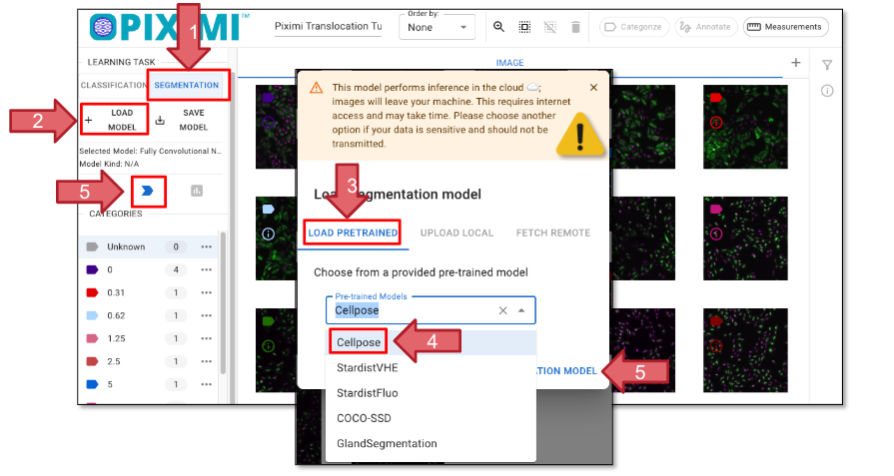
Fig. 42 Carregando um modelo de segmentação.#
Observe que as etapas anteriores foram executadas em sua máquina local, o que significa que suas imagens estão armazenadas localmente. No entanto, a inferência do Cellpose é executada na nuvem, o que significa que suas imagens serão enviadas para processamento. Se suas imagens forem altamente sensíveis, tenha cuidado ao usar serviços baseados em nuvem.
4. Visualize o resultado da segmentação e corrija os erros de segmentação#
🔴 PARA FAZER
Clique na aba CELLPOSE_CELLS para verificar as células individuais que foram segmentadas. Clique na aba “IMAGEM” e depois em “Anotar” para verificar a segmentação de toda a imagem.
Opcionalmente, aqui você pode refinar manualmente a segmentação usando as ferramentas do anotador. O anotador Piximi oferece diversas opções para adicionar, subtrair ou interseccionar anotações. Além disso, a ferramenta de seleção permite redimensionar ou excluir anotações específicas. Para começar a editar, selecione imagens específicas ou todas clicando na caixa de seleção na parte superior.
Opcionalmente, você pode ajustar os canais: embora existam dois canais neste experimento, o sinal dos núcleos é duplicado nos canais vermelho e verde. Este projeto foi projetado para ser compatível com daltonismo e produzir uma cor magenta para os núcleos. O canal verde também inclui sinais citoplasmáticos.
Outro motivo para duplicar os canais é que alguns modelos — como o modelo Cellpose que usamos hoje — exigem uma entrada de três canais.
Você pode optar por segmentar manualmente as células para gerar máscaras para dados de verdade básica.
Classificar células#
Motivo para isso: Queremos classificar as ‘CELLPOSE_CELLS’ com base na distribuição de GFP (em núcleos, citoplasma ou sem GFP) sem rotular todas elas manualmente. Para isso, podemos usar a função de classificação do Piximi, que nos permite treinar um classificador usando um pequeno subconjunto de dados rotulados e, em seguida, classificar automaticamente as células restantes.
🔴 PARA FAZER
Acesse a aba CELLPOSE_CELLS que exibe os objetos segmentados (seta 1, figura 26)
Clique na aba Classificação no painel esquerdo (seta 2, figura 26).
Crie novas categorias clicando em “+ Categoria”. Adicione as três categorias “Cytoplasmatic_GFP”, “Nuclear_GFP” e “No_GFP” (Seta 3, Figura 26).
Clique nas imagens que correspondem aos seus critérios. Você pode selecionar várias células pressionando Command (⌘) no Mac ou Shift no Linux. Tente atribuir ~20–40 células por categoria. Após selecionar, clique em “Categorizar” para atribuir os rótulos às células selecionadas.
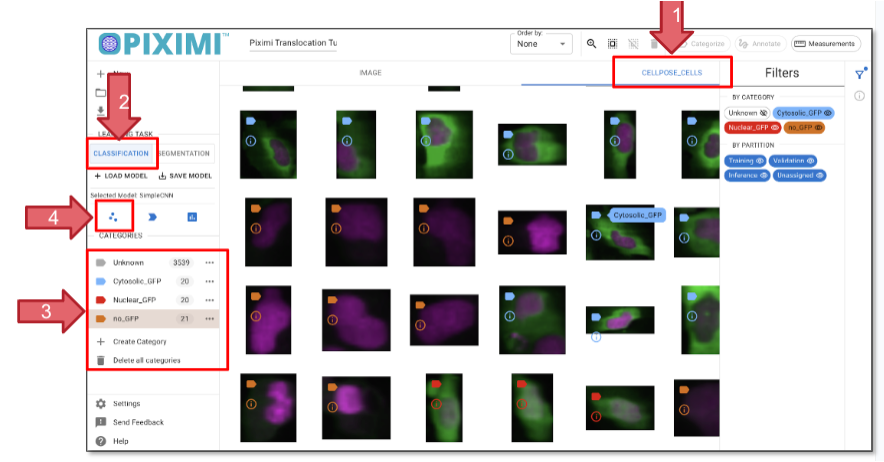
Fig. 43 Classificando células individuais com base na presença e localização de GFP.#
6. Treine o modelo do Classificador#
🔴 PARA FAZER
Clique no ícone “
 - Ajustar Modelo” para abrir as configurações de hiperparâmetros do modelo. Para o exercício de hoje, ajustaremos alguns parâmetros:
- Ajustar Modelo” para abrir as configurações de hiperparâmetros do modelo. Para o exercício de hoje, ajustaremos alguns parâmetros:Clique em “Configurações de arquitetura” e defina a arquitetura do modelo como SimpleCNN.
Atualize as dimensões de entrada para:
Linhas de entrada: 48
Colunas de entrada: 48
Canais: 3 (já que nossas imagens estão no formato RGB)
(Você pode mudar para outros números, como 64, 128)
Clique na aba “Configuração do Conjunto de Dados” e defina a Porcentagem de Treinamento como 0,75, o que reserva 25% dos dados rotulados para validação.
Ao clicar em “Ajustar Classificador” no Piximi, dois gráficos de treinamento aparecerão: “**Precisão vs. Épocas’’ e **”Perda vs. Épocas’’. Cada gráfico mostra curvas para os dados de treinamento e validação.
No gráfico de precisão, você verá o quão bem o modelo está aprendendo. Idealmente, a precisão tanto do treinamento quanto da validação deve aumentar e permanecer próxima.
No gráfico de perdas, valores menores significam melhor desempenho. Se a perda de validação começar a aumentar enquanto a perda de treinamento continua caindo, o modelo pode estar com sobreajuste.
Esses gráficos ajudam a entender como o modelo está aprendendo e se ajustes são necessários.
7. Avaliar modelo:#
🔴 A FAZER
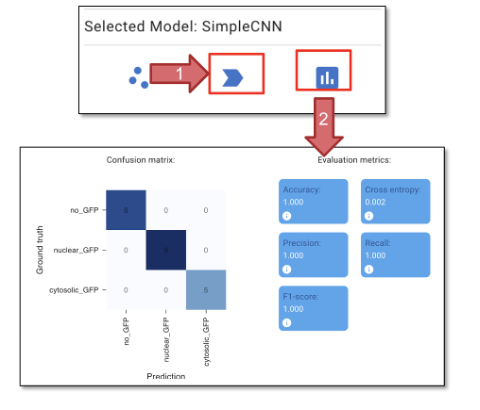
Fig. 44 Treinamento e validação do classificador.#
Clique em “Prever Modelo” (figura 28, seta 1) para aplicar o modelo que acabamos de treinar. Esta etapa gerará previsões nas células que não anotamos.
Você pode revisar as previsões na guia CELLPOSE_CELLS e excluir quaisquer categorias atribuídas incorretamente.
Opcionalmente, você pode continuar usando os rótulos para refinar a verdade básica e aprimorar o classificador. Esse processo faz parte da classificação humana no ciclo, na qual você corrige e treina o modelo iterativamente com base na entrada humana.
Clique em “Avaliar Modelo” (figura 28, seta 2) para avaliar o modelo que acabamos de treinar. As métricas de confusão e de avaliação podem ser comparadas com a verdade básica.
Clique em “Aceitar previsão (Manter)” para atribuir os rótulos previstos a todos os objetos.
8. Medição#
Assim que estiver satisfeito com a classificação, prosseguiremos com a medição dos objetos. O objetivo do exercício de hoje é determinar a concentração mínima de Wortmannin necessária para bloquear a exportação de FOXO1A-GFP dos núcleos. Para isso, podemos medir a intensidade total de GFP na imagem ou no objeto.
🔴 PARA FAZER
Clique em “Medição” no canto superior direito.
Clique em Tabelas (Seta 1), selecione Imagem e clique em “Confirmar” (Seta 2).
Selecione “MEDIÇÃO” no painel esquerdo. Observe que a etapa de medição pode levar algum tempo para ser processada.
Clique em “Categoria” para incluir todas as categorias na medição.
Em “Total”, clique em “Canal 1” (Seta 3) para selecionar a medição para GFP. Você verá a medição na aba “GRADE DE DADOS”. As medições são apresentadas como valores médios ou medianos, e o conjunto de dados completo está disponível ao exportar o arquivo .csv.
9. Visualização#
Após gerar as medições, você pode plotá-las.
🔴 PARA FAZER
Clique em “PLOTS” (Figura 30, Seta 1) para visualizar as medições.
Defina o tipo de gráfico como “Swarm” e escolha um tema de cores de acordo com sua preferência.
Selecione “Y-axis” como “intensity-total-channel-1” e defina “SwarmGroup” como “category”; isso gerará uma curva mostrando como a intensidade da GFP varia entre as diferentes categorias (Figura 30, Seta 2).
Selecionar “Show Statistics” exibirá a média, bem como os limites de confiança superior e inferior, no gráfico.
Opcionalmente, você pode experimentar diferentes tipos de gráfico e eixos para ver se os dados revelam insights adicionais.
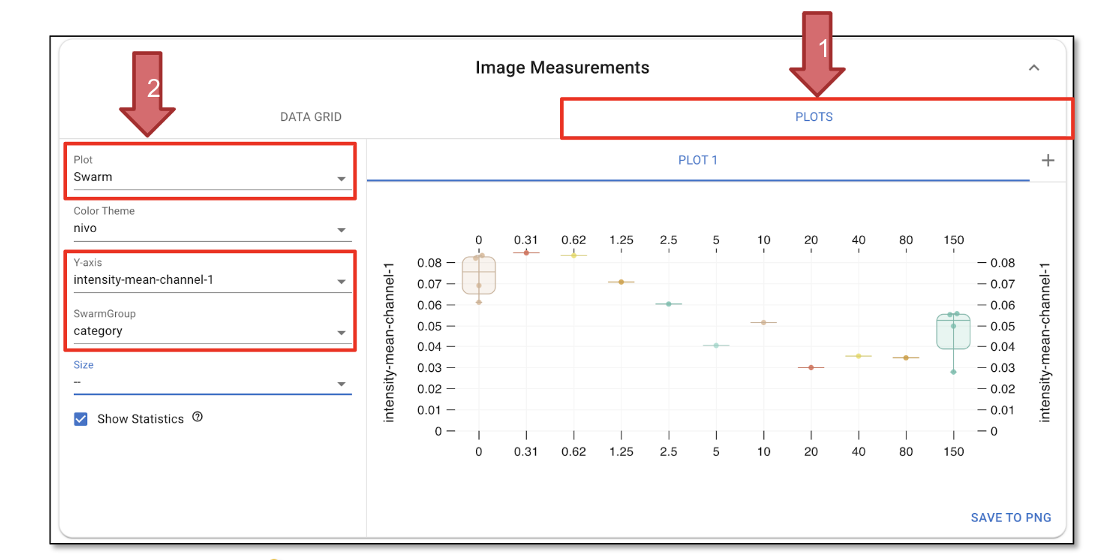
Fig. 45 Resultados do gráfico.#
10. Exportar resultados e salvar o projeto#
🔴 PARA FAZER
11. Informações de apoio#
Confira o artigo do Piximi: https://www.biorxiv.org/content/10.1101/2024.06.03.597232v2
Confira a documentação do Piximi:Documentação do Piximi:https://documentation.piximi.app/intro.html
Relatar bugs/erros ou solicitar recursos piximi/documentation#issues